サービス概要
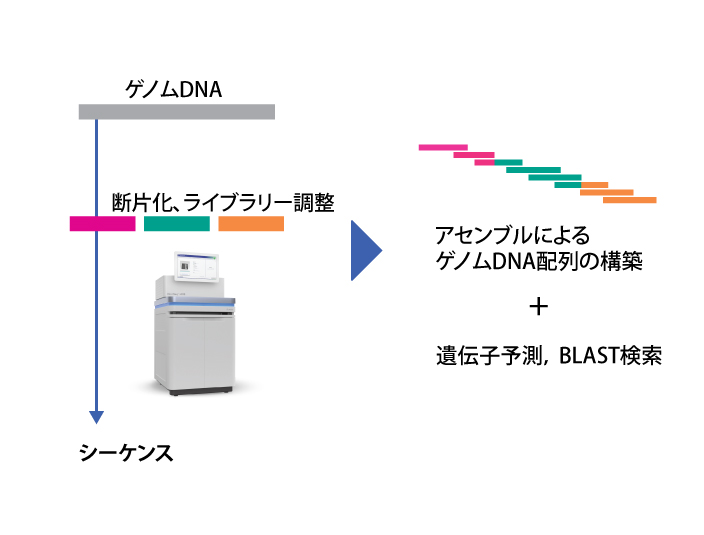
次世代シーケンサーを用いて、ゲノム配列が未知の生物種(リファレンス配列の無い生物種)についてゲノムDNAのシーケンスを行い、取得した配列情報を用いて新規ゲノム構築(de novo アセンブル)を行います。ホールゲノムシーケンス(WGS)とも呼ばれます。
推定ゲノムサイズに応じて、取得するデータ量を調整します。
また、シーケンスを行うための装置やライブラリの調製法を検討することで、目的や状況に応じた対応が可能です。
実施可能なアプリケーション例
ショットガン シーケンス(DNAシーケンス)
イルミナ社のショートリードのシーケンサー(HiSeq, NovaSeq6000など)を用いて、低コストに大量のデータを取得します。ゲノム解析を進める際の基本となるデータになります。
取得データ量の目安は、推定ゲノムサイズの100倍程度になります。
代表的な仕様
- ライブラリ調製:TruSeq DNA PCR-Free もしくは TruSeq DNA nano Kit
- シーケンス条件:NovaSeq6000, 150bp, paired end, 数十~数百 Gb /sample
- データ解析 :de novo アセンブル、遺伝子予測・BLAST検索(アノテーション付加)
メイトペアシーケンス
メイトペアシーケンスとは、ゲノム上で 数kb (3, 5, 8, 10kb) 離れた領域を含むDNA配列(ライブラリー)を調製し、その両端をシーケンスする方法です。
ゲノムDNAを数kbpのサイズで断片化し、その断片を環状化します。この環状化DNAを再度数百bpサイズで断片化すると、環状化前に端同士(メイトペア)だった配列が繋がった断片と、内側(インサート)の配列を持つ断片が得られます。
このメイトペアの断片を回収しペアエンドシーケンスすることで、数kb 離れた配列をメイトペアリードとして得ることが可能です。
結果として、両リードが数kbの距離関係にあることを決定できます。
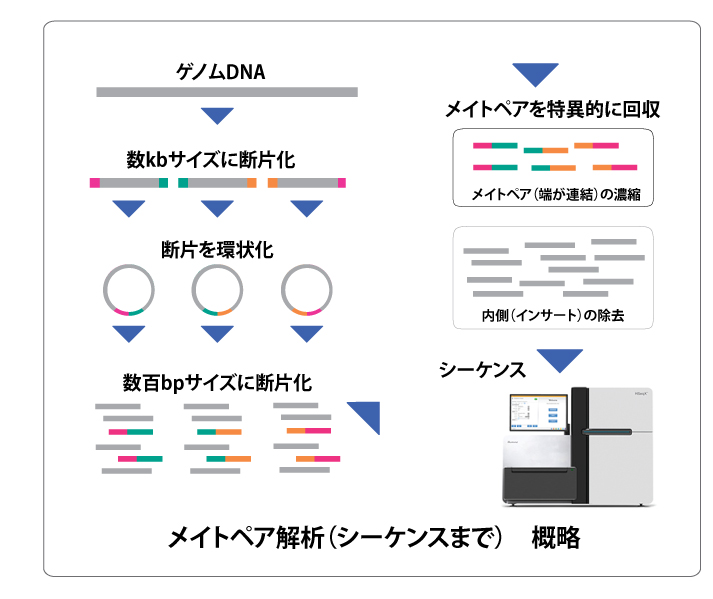
一般的にメイトペア解析を行う際には、同時に ショットガンシーケンス(DNAシーケンス)を実施します。
データ解析(新規ゲノム構築)を行う際に、まず、ショットガンシーケンスで得られたリードを用いて de novo アセンブル を実施してコンティグを構築します。
ここで構築した各コンティグは、どのようま位置関係にあるか不明な状態です。このコンティグ群に、メイトペアリードを当てることで、コンティグ間の位置関係や向きを整理してスキャホールドの作製が可能となります。
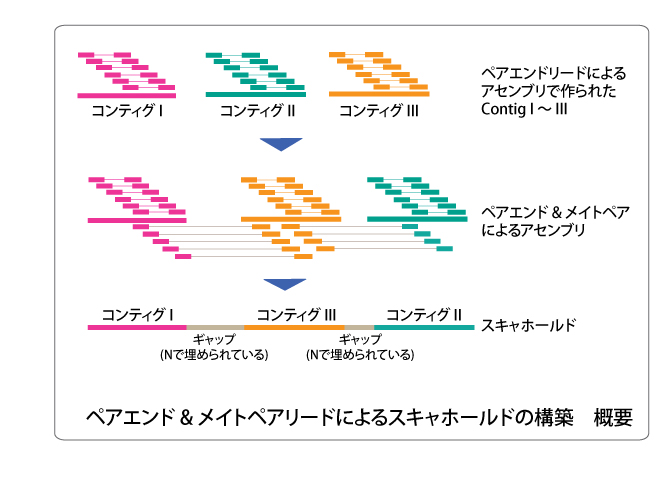
メイトペアリードは、真核生物など比較的ゲノムサイズの大きな生物のゲノム配列を構築する際には有効な方法になります。特に長さの異なる複数のメイトペアリードをアセンブルに用いることで、より長く、より正確なスキャホールドを得ることができます。
代表的な仕様
- ライブラリ調製:3 kb, 5 kb, 8 kb (3種類)
- シーケンス条件:NovaSeq6000, 150bp, paired end, 数十~数百 Gb /sample
- データ解析 :de novo アセンブル
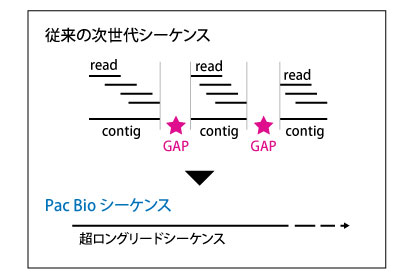
PacBio シーケンス(数十kb のロングリードシーケンス)
数十kb のロングリードの取得が可能なPacBio シーケンスを利用することで、コンティグが少なく、ギャップが生まれにくい、完全長ゲノムDNAの構築を目指す手法になります。
取得データ量の目安は、100倍程度になりますので、推定ゲノムサイズが数Mb程度のバクテリアのゲノムシーケンスでは、RSII の使用がお勧めになります。ゲノムサイズの大きい生物種になりますと、Sequel、 Sequel II の利用をお勧めしております。
■取得データ量の目安
PacBio RSII :約0.5~1Gb/Cell
PacBio Sequel : 約4Gb程度/Cell
PacBio Sequel II :約50~100Gb程度/Cell
代表的な仕様
- シーケンス条件:PacBio RSII, Sequel, Sequel II
- データ解析 :de novo アセンブル, ギャップクロージング
