
サービス概要
RNA-Seq(RNAシーケンス)は、次世代シーケンスを用いて取得したリードの情報(生データ)をデータ解析することで、遺伝子の発現量が解析できる手法です。
リファレンス配列のある生物種だけでなく、リファレンス配列のない生物種においても遺伝子発現解析の実施が可能です。このように配列未知の生物種に対してもアプローチが可能である点は、既存の手法であるマイクロアレイとは大きく異なる点になります。
また、一般的な手法では、total RNA のご提供を頂き、polyA を利用して mRNA を回収してサンプル調製を進めますが、polyA が無い生物種である場合には、リボソーマルRNAを除去する手法(リボゼロ処理)を利用してサンプル調製を進めます。

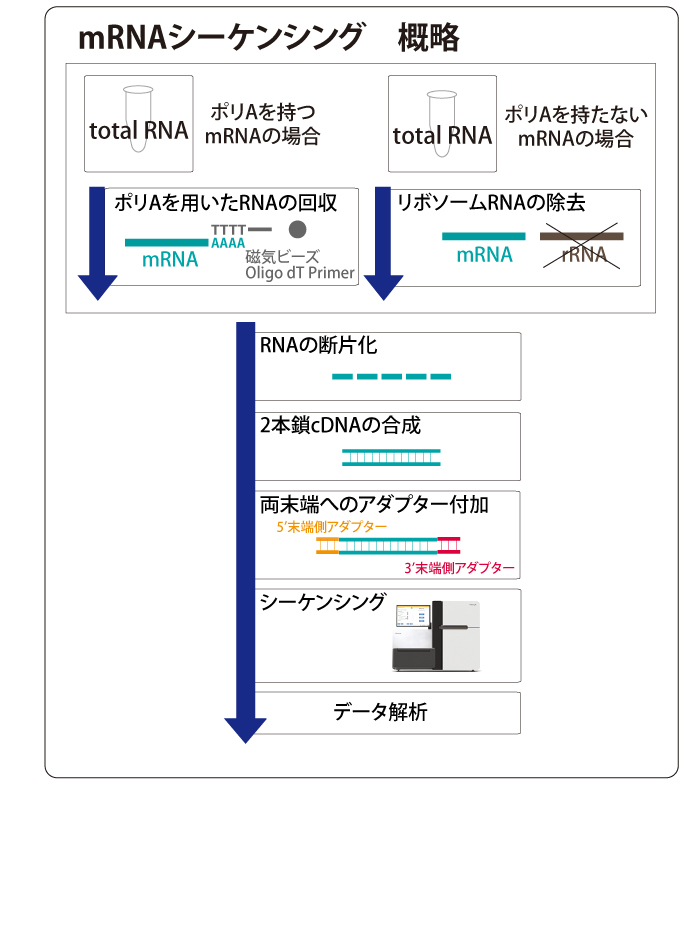
上記のように、シーケンスを行うためのサンプル前処理法やシーケンスに用いる装置、データ解析の手法を最適化することで、下記のような様々なアプリケーションにも対応が可能です。
- 微生物のRNA-Seq(リボゼロ処理)
- total RNA-Seq(リボセロ処理)
- 微量サンプルの RNA-Seq(SMARTerによる増幅)
- Iso-Seq(PacBioを利用)
- small RNA-Seq(miRNA-Seq)
仕様
RNA-Seq(PolyA による回収)
<代表的な仕様>
- ご提供サンプル:total RNA (1 microG 以上)
- ライブラリ調製:TruSeq Stranded mRNA Library Prep(PolyA による回収)
- シーケンス条件:NovaSeq X Plus, 4 Gb/サンプル, 100 bp, paired end
- データ解析:リファレンス配列に対するマッピング、発現値算出、発現比較
<内容>
ヒト・マウスなどのpolyAを持つ mRNA を対象とした解析になります。
リファレンス配列の無い生物種である場合、得られたリードをde novo アセンブル・BLAST検索することで、リファレンス配列を作成して解析を進めることが可能です。
微生物のRNA-Seq(リボゼロ処理)
<代表的な仕様>
- ご提供サンプル:total RNA (1 microG 以上)
- ライブラリ調製:TruSeq stranded mRNA Library(bacteria)
- シーケンス条件:NovaSeq X Plus, 4 Gb/サンプル, 100 bp, paired end
- データ解析:リファレンス配列に対するマッピング、発現値算出、発現比較
<内容>
polyAの無いmRNAを持つ微生物のRNA-Seqを実施する場合は、通常の手法ではサンプル調製ができないため、リボソーマルRNAを除去する手法でライブラリ調製を行います。
total RNA-Seq(リボセロ処理)
<代表的な仕様>
- ご提供サンプル:total RNA (1 microG 以上)
- ライブラリ調製:TruSeq Stranded total RNA Library Prep Kit with RiboZero kit
- シーケンス条件:NovaSeq X Plus, 10 Gb/サンプル, 100 bp, paired end
- データ解析:リファレンス配列に対するマッピング、発現値算出、発現比較
<内容>
ヒトやマウス由来のサンプルであっても、non-coding RNAやlncRNAと呼ばれるpolyAの無いRNAを対象にした解析を希望される場合に用いる手法です。Total RNA からリボソーマル RNA を除去してライブラリ調製を進めます。
10 Gb/sample 程度のデータを取得することをお勧めしております。
微量サンプルの RNA-Seq(SMARTerによる増幅)
<代表的な仕様>
- ご提供サンプル:total RNA (0.01 ug 以上)
- ライブラリ調製:SMARTer Ultra low RNA Kit + TruSeq library construction
- シーケンス条件:NovaSeq X Plus, 4 Gb/サンプル, 100 bp, paired end
- データ解析:リファレンス配列に対するマッピング、発現値算出、発現比較
<内容>
微量サンプルの場合には、SMARTer を用いた増幅を含む仕様でライブラリ調製を進めることが可能です。10 ng 程度の微量なサンプルからでもデータを取得できる可能性があります。
ただ、こちらのSMRTer による増幅は、結果をお約束してお受けすることが出来ません。
万が一、良好な結果が得られない場合でも費用が必要になりますので、ご理解を頂いた上で、ご検討をお願いします。
Iso-Seq(PacBioを利用)
<代表的な仕様>
- ご提供サンプル:total RNA (1.2 microG 以上)
- ライブラリ調製:Sequel II Iso-Seq Library Construction
- シーケンス条件:PacBio Sequel II 1cell
- データ解析:isoform Profiling(リファレンスあり)
※ HiFiでの対応となります。
※ 1cellでHi-Fiリードで 3-5Mリード 程度のデータ量が見込めます。
<内容>
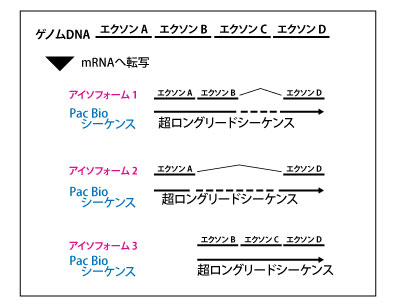
スプライシングバリアントの解析が目的である場合には、数十kbのロングリードのシーケンスが可能なPacBioを用いた Iso-Seq の対応が可能です。
Illumina社に代表されるショートリードのシーケンサーでは、アイソフォーム(スプライシングバリアント)の判別が困難です。一方、ロングリードのPacBioシーケンスでは、1つのリードで mRNA 分子全体を網羅することができる(mRNAの全長を 1リードでシーケンスできる)ため、多様なアイソフォームが混在する場合でも、それらを見分けることができ、アイソフォームごとの発現解析を実施することが可能です。
small RNA-Seq(miRNA-Seq)
<代表的な仕様>
- ご提供サンプル:total RNA(3microG 以上)
- ライブラリ調製:NEBNext Small RNA Library
- シーケンス条件:NovaSeq X Plus, 0,5 Gb/サンプル, 50 bp, single end
- データ解析:あり(発現値算出、発現量比較)
※ 150bpでシーケンス後、50SEにトリミングして納品します。
<内容>
18~30塩基程度の small RNA の両端にアダプターを付加し、シーケンスを行うことで、small RNA の同定と発現量を解析するサービスです。
検出できるダイナミックレンジが広いので、 miRNA などの small RNA の発現量を調べたい場合に有効です。
納期
目安の納期は、サンプル受領から品質チェックの結果のご報告までが 1~2 週間程度、
品質チェックの通過からシーケンスの完了までが 6~8 週間程度、データ解析が2 週間程度の見込みです。
サンプル受領のタイミングや取得したデータによっては、納期が前後する場合がありますので、ご了承ください。
